データ解析のための統計モデリング入門(通称、緑本)を読み始めました。
述べられている理論を整理しつつ、Rでの実装をPythonに置き換えた際のポイントなども深掘りしていきます。
今回は第3章です(前回の投稿はこちら)。実装は以下で公開しています。
3 一般化線形モデル(GLM)
個体ごとに異なる説明変数によって応答変数が変化する統計モデルとしてポアソン回帰を導入する。ポアソン回帰は一般線形化モデル(GLM)の一つです。
この章では2つのデータを使います。
1つは、第3章で扱われている架空植物100個体の種子数のデータで、著者サイトから入手できます。目的変数は種子数(y)です。
http://hosho.ees.hokudai.ac.jp/~kubo/ce/IwanamiBook.html
もう1つは、前回の投稿でも使いました、UCIで提供されているStudent Performance Data Setです。目的変数は欠席数(absences)です(前回同様、外れ値の除去や、欠席2回を1回とカウントするなど、若干の前処理を行っています)。
https://archive.ics.uci.edu/ml/datasets/student+performance
この投稿では種子数データ、欠席数データとそれぞれ呼ぶことにします。
3.1 例題: 個体ごとに平均種子数が異なる場合
種子数データでは、以下の個体差がある架空の植物100個体を想定している。個体差はいずれも観測データとして与えられている。平均種子数に影響を与えているのかについて知りたい。
- 体サイズ
- 施肥処理の有無(50個体は施肥処理有り)

また、欠席数データでは、以下の特徴(個体差)を持つ生徒389人に対して、欠席数(absences)との関係を調べます。
- 年齢 (age)
- 恋人がいるかどうか (romantic)

3.2 観測されたデータの概要を調べる
Pandasでデータの概要を見る場合は、head()で先頭5行、info()でデータの型、value_counts()で値の分布、describe()で要約統計量をそれぞれ出力すると良いかと思います。
# 先頭5行の表示 data.head() # 各列の型 data.info() # fの値の分布 data['f'].value_counts() # 数値列(x列とy列)の要約統計量 data.describe()
3.3 統計モデリングの前にデータを図示する
散布図と箱ひげ図でデータの分布を視覚化します。
種子数データでは、散布図では体サイズが大きくなるほど種子数も増える傾向が見られますが、箱ひげ図からは施肥処理による種子数の変化は読み取れません。
# 散布図 plt.scatter(data[data['f']=='C']['x'], data[data['f']=='C']['y'], marker='o', label='C') plt.scatter(data[data['f']=='T']['x'], data[data['f']=='T']['y'], marker='x', label='T') plt.legend(loc='best') plt.show()

# 箱ひげ図 fig, ax = plt.subplots() ax.boxplot([data[data['f']=='C']['y'], data[data['f']=='T']['y']]) ax.set_xticklabels(['C', 'T']) plt.show()

次に欠席数データについて見てみます。
散布図では、17歳をピークとした山になっていますが、概ね年齢が上がると欠席数も増える傾向がありそうです。箱ひげ図では、恋人がいる方が欠席数は多くなることが伺えます。


3.4 ポアソン回帰の統計モデル
個体iの種子数 がポアソン分布に従っていると仮定します。
個体ごとに異なる平均 を説明変数(共変量)
の関数として、仮に以下で定義する。
はパラメータ(係数)と呼び、当てはまりの良い値を選ぶ。
- 右辺
は、線形予測子
- 左辺
の関数は、リンク関数
- 対数(
)の場合は、対数リンク関数と呼ばれ、ポアソン分布に使われる
- 対数リンク関数、ロジットリンク関数(ロジスティック回帰で用いる)は、正準リンク関数と呼ばれる
- 対数(
当てはまりの良さは対数尤度関数で定量化し、対数尤度が最大となる を求めていましたが、それを今回導入したパラメータの関数に置き換えます。以下の関数を最大にする
を求めます。
最尤推定量は、Rだと> fit <- glm(y ~ x, data = d, family = poisson)で求められますが、PythonではstatsmodelやPyStanでこうした計算が簡単に行なえます。今回はstatsmodelを使います。
http://www.statsmodels.org/stable/index.html
以下のコードではいずれも最尤推定を計算しています。実行前にstatsmodelsをインストール( pip install statsmodels )する必要があります。
statsmodelsには2種類のAPI(statsmodels.apiとstatsmodels.formula.api)があります。statsmodels.apiでは説明変数と目的変数を受け取るピュアな関数が提供されます。一方、statsmodels.formula.apiでは目的変数と説明変数の関係式をRのフォーマットで渡せます。
import statsmodels.api as sm # 最尤推定(切片も含む場合はadd_constant()で値を渡す) results = sm.Poisson(data['y'], sm.add_constant(data['x'])).fit() #results = sm.GLM(data['y'], sm.add_constant(data['x']), family=sm.families.Poisson()).fit() # 結果の出力 results.summary()
import statsmodels.formula.api as smf # 最尤推定 results = smf.poisson('y ~ x', data=data).fit() # 結果の出力 results.summary()
結果は以下のように出力されます。
- 最尤推定値(coef)はそれぞれ、
(Interceptまたはconst)は1.2917、
(x)は0.0757
- このときの最大対数尤度(Log-Likelihood)は-235.39

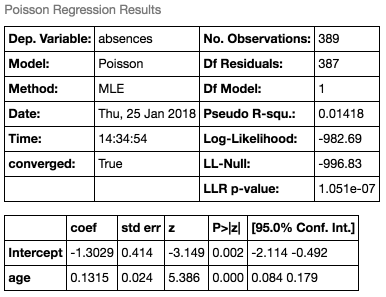
同様に、欠席数データに対しても適用すると、以下の結果になります。
最尤推定値(coef)は、 (Intercept)は-1.3029、
(age)は0.1315で、このときの最大対数尤度は-982.69です。第2章のモデルの最大対数尤度は-996.83(
の時)だったので、それよりも当てはまりの良いモデルとなっていることがわかります(ちなみに
が一定のモデルの最大対数尤度はLL-Nullの値と一致します。第4章で詳しく見ていきます)。
3.5 説明変数が因子型の統計モデル
施肥処理の有無を表す のように、Rでは数量型以外で特定の値(水準)をとる変数を因子型と言います。因子型の場合は、ダミー変数に置き換えてモデル化します。
施肥処理の効果のみを考慮した以下のモデルでは、 がダミー変数ですが、
の時は
、
の時は
となるようにします。
pandasではget_dummies()でダミー変数に置き換えることができます。
# f=Tの場合に1となる列d(ダミー変数)を追加 data['d'] = pd.get_dummies(data['f'])['T']
結果は以下のとおりです。
0か1しか値を持たないにもかかわらず、最尤推定値は0.0128と小さな値となっていることから、種子数に対する影響は小さいことがわかります。

欠席数データに対しても、恋人の有無をダミー変数に置き換えたモデルを作りました。
こちらは、最尤推定値が0.2250でそれなりに影響力はありそうです。

3.6 説明変数が数量型+因子型の統計モデル
複数の説明変数を組み合わせる場合は、線形予測子で和として表現する。
Pythonでも単純で、'y ~ x+d'と表現できます。
results = smf.poisson('y ~ x+d', data=data).fit()
種子数データの結果を見ると、fはマイナスの推定値となっており、種子数については逆効果となりそうです。 一方、最大対数尤度は少し良くなっています。

得られた最尤推定値を当てはめると、 は以下の式で導出されます。logが取り払われたことで、乗算で
に影響を与えることがわかります。
欠席数データでは、 は以下の式になります。2変数となったことで、最大対数尤度も-979.35と良くなっています。

3.7 「何でも正規分布」「何でも直線」には無理がある
確率分布を等分散の正規分布、リンク関数を恒等リンク関数(何もしない、つまり で表現される
)にすると、線形モデル(LM)と呼ばれる。
そのLMの1つが直線回帰です。直線回帰は以下の特徴を持つため、今回のカウントデータでは不適切といえる。
応答変数にあわせて、確率分布は適切に選ぶべき。
