データ解析のための統計モデリング入門(通称、緑本)を読み進めています。
述べられている理論を整理しつつ、Rでの実装をPythonに置き換えた際のポイントなども深掘りしていきます。
今回は第9章です。PyMC3を使って、GLMをベイズモデルで表現します。実装は以下で公開しています。
9 GLMのベイズモデル化と事後分布の推定
9.1 例題:種子数のポアソン回帰(個体差なし)
架空植物20個体において体サイズと種子数を計測した。個体iにおける体サイズを 、種子数を
とします。得られたデータから体サイズ
と種子数
の関係について調べる、というのが今回の例題です。
計測結果は以下にプロットしています。
なお著者サイトで提供されているサンプルデータはRDataですので、Pythonで扱えるようにするため、PypeRでロード後にDataFrame化しています。PypeRについては以前の投稿も参考にしてください。

種子数は上限が無い離散値ですので、ポアソン分布でばらつきを表現できそうです。
statsmodelsで最尤推定すると、ポアソン分布の平均 は下式で得られます(なお、このデータは作成するときには
から生成されたとのことです)。
$$ \lambda_i = \exp(1.5661 + 0.0833x_i) $$
import statsmodels.formula.api as smf result = smf.poisson('y ~ x', data=data).fit() result.summary()

9.2 GLMのベイズモデル化
ベイズモデル化したとしても、中核はポアソン回帰のGLM。
- 平均
のポアソン分布
に従う
- 線形予測子と対数リンクを使い、
で指定する
このモデルでの尤度関数Lは以下となります。 を定数としている(Lのパラメータになっていない)ことに注意してください。
$$ L(\beta_1, \beta_2)=\prod_i p(y_i \mid \lambda_i)=\prod_i p(y_i \mid \beta_1, \beta_2, x_i) $$
ある において
(
)が得られる確率は、尤度関数と一致します(この関係は8.4で導入・説明しています)。
$$ p({\bf Y} \mid \beta_1, \beta_2)=L(\beta_1, \beta_2) $$
ベイズモデルの事後分布は、尤度×事前分布に比例するので、以下の関係が成り立ちます(こちらも8.4を参考にしてください)。
$$ p(\beta_1, \beta_2 \mid {\bf Y}) \propto p({\bf Y} \mid \beta_1, \beta_2)p(\beta_1)p(\beta_2) $$
9.3 無情報事前分布
まずは事前分布 と
(まとめて
)を設定する。
データ が得られていない状態で決める事前分布なので、無情報事前分布と呼ばれます。つまり、どんな値(
] )でも良いのです。
こうした分布はの生成方法は2つあります。
- 広い範囲 (例えば[-100000, +100000])の一様分布
- 平均0で標準偏差が大きい(平べったい)正規分布
分散100の正規分布は以下のようにプロットできます。今回は標準偏差100の正規分布を、事前分として使います。

9.4 ベイズ統計モデルの事後分布の推定
事前分布が定まったので、事後分布 をMCMCサンプリングで推定します。
書籍では、WinBUGS+R2WinBUGSで、RからMCMCサンプリンによる推定が行われています。
ベイズモデルでパラメータ推定できるPythonパッケージとして、PyStanやPyMCが有名です。今回はPyMC3を使います。いつも通り、pip install pymc3でインストールしておきます。
PyMC3によるGLMのパラメータ推定は、3ステップで行います。
- モデルを定義する
- サンプリングして推定する
- 得られた結果を確認する
import pymc3 as pm # モデルを定義する with pymc3.Model() as model: # 事前分布をN(0, 100)の正規分布で設定 beta1 = pymc3.Normal('beta1', mu=0, sd=100) beta2 = pymc3.Normal('beta2', mu=0, sd=100) # 線形予測子θをβ1+β2xで設定 theta = beta1 + beta2*data['x'].values # ログリンク関数(log(μ)=θ⇔μ=exp(θ))を設定し、ポアソン分布で推定する y = pymc3.Poisson('y', mu=np.exp(theta), observed=data['y'].values) # サンプリングして推定する with model: # 101個目から3個置きでサンプルを取得するチェインを3つ作る # NUTSではburnとthinが効いていない? trace = pymc3.sample(1600, burn=100, thin=100, njobs=3, random_seed=0) # 得られた結果を確認する pymc3.traceplot(trace) # サンプリング過程を表示する pymc3.summary(trace) # 推定結果を表示する
モデルを定義する
まずwith句でモデルクラス(pymc3.Model)を作成し、事前分布、線形予測子、リンク関数、確率分布を指定します。
の事前分布には、それぞれ平均0・標準偏差100の正規分布(pymc3.Normal)を使う
- 線形予測子は
- ポアソン分布(pymc3.Poisson)で尤度を計算
- ログリンク関数
で平均
を設定
- ログリンク関数
import pymc3 # モデルを定義する with pymc3.Model() as model: # 事前分布をN(0, 100)の正規分布で設定 beta1 = pymc3.Normal('beta1', mu=0, sd=100) beta2 = pymc3.Normal('beta2', mu=0, sd=100) # 線形予測子θをβ1+β2xで設定 theta = beta1 + beta2*data['x'].values # ログリンク関数(log(μ)=θ⇔μ=exp(θ))を設定し、ポアソン分布で推定する y = pymc3.Poisson('y', mu=np.exp(theta), observed=data['y'].values)
サンプリングしてパラメータ推定する
次に、pymc3.sampleメソッドでサンプラーの定義とサンプリングを行い、パラメータを推定する。
- 最初の引数(draws)で、サンプル数を指定
- stepで、サンプリングアルゴリズムを指定
- Metropolis、HamiltonianMC、NUTSなどが選択できます(デフォルトはNUTS)
- tuneで、先頭から捨てるサンプル数を指定する(WinBUGSではn.burninで指定する値)
- サンプルの最初の方は、ランダムに選ばれた初期値の影響を大きく受けるため、捨てた方が良い
- njobsで、チェイン数(サンプル列数)を指定
- 3を指定すると、3つの異なる初期値からそれぞれサンプリングが行われる
- observedに、観測された従属変数(ここではy)を渡します
- 2個飛ばしでサンプリング(つまり合計500個)するために、スライス表記[::3]でサンプリング過程を取得
# ハミルトニアンモンテカルロ法
with model:
# 101個目から3個置きでサンプルを取得するチェインを3つ作る
trace = pymc3.sample(1500, step=pymc3.HamiltonianMC(), tune=100, njobs=3, random_seed=0)[::3]
得られたサンプリング過程(trace)は、添字でアクセスできます。
print('Trace type:', type(trace)) # Trace type: <class 'pymc3.backends.base.MultiTrace'> print('Trace length:', len(trace)) # Trace length: 500 print('trace[0]:', trace[0]) # trace[0]: {'beta1': 2.0772965015391716, 'beta2': -0.02971672503615687}
得られた結果を確認する
サンプリング後には、初期値やサンプリング数などの設定値が適切であったかどうかを確認する必要があります。
pymc3.traceplotメソッドで、サンプリング過程がグラフ化できます。
pymc3.traceplot(trace)
メトロポリス法(上図)とハミルトニアンモンテカルロ法(下図、以降はHMCと表記)で並べて比較してみます。
- メトロポリス法については、以前の投稿を見てください
- HMCについては、こちらの方の記事と、リンクされている伊庭さんのPDFが役に立つかと思います (まだちゃんと調べられてないです)
右図のパラメータの推移を見ると、HMCではサンプル列同士が近づきつつありますがまだ不安定な状態にある(つまりサンプリング数が不足している)ことがわかります。対して、HMCではいずれのサンプル列同士も十分近づき、類似した波形となっています。
いずれも、3つのサンプル列がプロットされていることや、サンプル数が500(1500から2つ飛ばしでサンプリングしているため)となっていることに注意してください。
メトロポリス法

HMC

また、pymc3.summaryメソッドで、各パラメータの推定値とそれぞれの統計値を確認できます。
pymc3.summary(trace)
こちらもメトロポリス法(上表)とHMC(下表)で並べてみます。
各項目の詳細は次節で見ていきますが、このうちRhatで表記されている 指数はサンプル列間のばらつきを表す値で、パラメータ毎に求められます。この値が1に近いほどサンプル列間のばらつきよりも列内のばらつきが大きくなるので、収束していると言えます。経験的には
が1つの目安ですが、
も十分なサンプル数でない場合は安定した結果が得られないので注意が必要です。
メトロポリス法では1.007、HMCでは0.999なので、後者の方が僅かですがより収束していると言えそうです。
メトロポリス法
HMC

9.5 MCMCサンプルから事後分布を推定
得られた推定結果から事後分布を確認します。
HMCで得られたサンプリング過程を再掲します。
左図は、 と
の周辺事後分布で、カーネル密度推定で近似された確率密度関数で表現されています。周辺事後分布は、あるパラメータ1つに関する事後分布で、ここでは
と
となります。

周辺事後分布に対して、 は同時事後分布と呼ばれます。
パラメータの組み合わせを散布図にプロットします。 と
の相関がかなり強いサンプリングが行われていることがわかります。本書の傾向と大きく異なっており、サンプリングアルゴリズム(本書ではWinBUGSのギブスサンプリング実装)の違いによるものと思われます。

なお、サンプリングした値はtrace(MultiTraceクラス)からget_valuesメソッド(返り値の型はnumpy.ndarray)で取得できます。
- 1番目の引数(varname)にパラメータ名を指定
- chainsオプションに、サンプル列のインデックスを渡す
# サンプル列数分だけ繰り返す for i in trace.chains: # 各サンプル列のパラメータの平均値を計算 beta1_averages += trace.get_values('beta1', chains=i) / trace.nchains beta2_averages += trace.get_values('beta2', chains=i) / trace.nchains
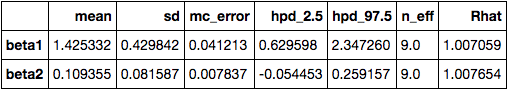
次に、HMCの統計量から事後分布の推定値を確認します。
- パラメータ
は平均1.5599で、95%信用区間は0.8630〜2.2622
は95%の事後確率で0.8630〜2.2622に収まる、と解釈できる
- パラメータ
は平均0.0826で、95%信用区間は0.0017〜0.2145
- n_effは有効なサンプルサイズで、サンプル間の相関が高いと、この値が小さくなります

9.6 複数パラメーターのMCMCサンプリング
8章では、1パラメータについてメトロポリス法でMCMCサンプリングする方法を説明しました。
メトロポリス法には、更新前と更新後の値の相関が強く、なかなか収束しないという問題がありました。
この問題を解決するサンプリング方法の1つにギブスサンプリングがあります。ギブスサンプリングは、「新しい値の確率分布を作ってその確率分布からランダムに選択する」という方法で値を更新することで、更新前後の値の相関を弱くします。
さらに今回の例題のように、複数のパラメータ( )のMCMCサンプリングを考える必要があります。
こうした場合、全てのパラメータを同時に更新するよりも、 と
を交互に少しずつ更新していく方が簡単です。
2パラメータのギブスサンプリングをまとめると以下のアルゴリズムとなります。
- (1)
の適当な初期値を設定する
- 例えば
と置く
- 例えば
- (2)
に従う乱数を発生させ、得られた値を新しい
とする
- 他の変量を全て定数とする一変量確率分布で、全条件付き分布(FCD)と呼ばれる
- FCDに従う乱数1つを発生させる(
が得られたとする)
- FCDからのサンプリングする方法は理解できませんでした
$$ p(\beta_1 \mid {\bf Y}, \beta_2=0.0) \propto \prod_i \frac{\lambda_i^{y_i} \exp(-\lambda_i)}{y_i !}p(\beta_1) $$
- (3)
に従う乱数を発生させ、得られた値を新しい
とする (2の逆)
$$ p(\beta_1 \mid {\bf Y}, \beta_1=2.052) \propto \prod_i \frac{\lambda_i^{y_i} \exp(-\lambda_i)}{y_i !}p(\beta_2) $$
- (4) この新しい
を記録する
- (5) 十分なサンプル数が得られるまで(2)〜(4)を繰り返す










