今回は不均衡なクラス分類で便利なimbalanced-learnを使って、クレジットカードの不正利用を判定します。
データセット
今回はkaggleで提供されているCredit Card Fraud Detectionデータセットを使います。
ヨーロッパの人が持つカードで、2013年9月の2日間の取引を記録したデータセットです。
1取引1レコードとなっており、各レコードには不正利用か否かを表す値(1ならば不正利用)を持っていますが、当然ながらほとんどが0で、極めて不均衡なデータセットとなっています。
また、個人情報に関わるため、タイムスタンプと金額以外の項目が主成分分析(および標準化)済みとなっていることも特徴です。
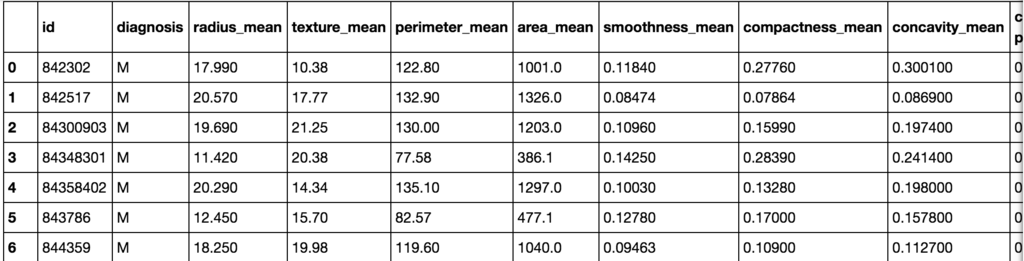
# ライブラリのインポート import pandas as pd from sklearn.model_selection import train_test_split from sklearn.metrics import confusion_matrix from sklearn.ensemble import RandomForestClassifier # CSVファイルをDataFrameへロード original_df = pd.read_csv('creditcard.csv') original_df.head()

不正利用かどうかは'Class'列に入っており、1ならば不正利用です。 Classが0のサンプル数が284,315に対して、1のサンプル数は492と、不均衡な分布です。
original_df['Class'].value_counts() # 0 284315 # 1 492
前処理などを行わず(ただしタイムスタンプ(‘Time’)は特徴量から削除します)、この段階でランダムフォレスト(決定木は100個)で学習・テストさせます。
結果、テストデータのスコアは99.95%という高い値となりましたが、もともとかなり偏りがあって全て0と答えるだけでも99.82%が達成されることを鑑みると、決して良い結果ではありません。 混合行列を見ても、不正利用であると誤って判定(偽陽性、False Positive: FP)しているサンプル数が10に対して、不正利用ではないと誤って判定(偽陰性、False Negative: FN)しているサンプル数が66になっており、不正利用ではないと判定されやすくなっていることがわかります。
# 説明変数Xと目的変数yの抽出 X = original_df.loc[:, 'V1':'Amount'] y = original_df.loc[:, 'Class':] # 学習データとテストデータの分離 X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.5, random_state=0) # ランダムフォレストによる学習 rfc = RandomForestClassifier(random_state=0, n_estimators=100) rfc.fit(X_train, y_train) # 検証 print('Train score: {:.4f}'.format(rfc.score(X_train, y_train))) print('Test score: {:.4f}'.format(rfc.score(X_test, y_test))) print('Confusion matrix:\n{}'.format(confusion_matrix(y_test, rfc.predict(X_test)))) print('f1 score: {:.4f}'.format(f1_score(y_test, rfc.predict(X_test)))) # Train score: 1.0000 # Test score: 0.9995 # Confusion matrix: # [[142151 10] # [ 66 177]] # f1 score: 0.8233
imblanced-learn
偏りが大きいデータセットに対して、多少の偽陽性が増えたとしても、偽陰性を減らしたい場合があります。 ここでは、誤検出が少し増えても不正利用を検出したい、というニーズです。
そうした場合に、学習に使われる陽性サンプルの割合を増やすことで偽陰性を減らす、という方法がしばしば採られるようです(当然、偽陽性は増えてしまいます)。 割合を操作するには、大きく括ると3つのやり方があります。
- 陰性サンプルを減らす(under-sampling)
- 陽性サンプルを増やす(over-sampling)
- 上記両方を行う
Pythonでは、imbalanced-learnを使うことで、こうしたサンプル数の操作を簡単にできます。
pip install -U imbalanced-learnでレポジトリからインストールできます。
しかし、PyPIで提供されているバージョンは0.2.1(2017/8/12現在)で、細かいパラメータ設定などができないため、開発バージョンを取得してインストールします。
やり方はここに記載のとおりです。
git clone https://github.com/scikit-learn-contrib/imbalanced-learn.git cd imbalanced-learn python setup.py install
under-sampling
まずは、under-samplingを行います。
imbalanced-learnで提供されているRandomUnderSamplerで、陰性サンプル(ここでは不正利用ではない多数派のサンプル)をランダムに減らし、陽性サンプル(不正利用である少数派のサンプル)の割合を10%まで上げます。
同じパラメータでランダムフォレストを使って学習・テストすると、FNが66から43まで減っていることがわかります。 一方でFPは10から157となっており、誤検出が大幅に増えています。
from imblearn.under_sampling import RandomUnderSampler # 不正利用のサンプル数をカウント positive_count_train = y_train['Class'].sum() print('positive count: {}'.format(positive_count_train)) # positive count: 249 # ランダムにunder-sampling rus = RandomUnderSampler(ratio={0:positive_count_train*9, 1:positive_count_train}, random_state=0) X_train_resampled, y_train_resampled = rus.fit_sample(X_train, y_train) print('X_train_resampled.shape: {}, y_train_resampled: {}'.format(X_train_resampled.shape, y_train_resampled.shape)) print('y_train_resample:\n{}'.format(pd.Series(y_train_resampled).value_counts())) # X_train_resampled.shape: (2490, 29), y_train_resampled: (2490,) # y_train_resample: # 0 2241 # 1 249 # dtype: int64 # ランダムフォレストにて学習 rfc = RandomForestClassifier(random_state=0) rfc.fit(X_train_resampled, y_train_resampled) # 検証 print('Train score: {:.4f}'.format(rfc.score(X_train_resampled, y_train_resampled))) print('Test score: {:.4f}'.format(rfc.score(X_test, y_test))) print('Confusion matrix:\n{}'.format(confusion_matrix(y_test, rfc.predict(X_test)))) print('f1 score: {:.4f}'.format(f1_score(y_test, rfc.predict(X_test)))) # Train score: 0.9968 # Test score: 0.9986 # Confusion matrix: # [[142004 157] # [ 43 200]] # f1 score: 0.6667
over-sampling
次にRandomOverSamplerを使って、over-samplingを行います。 under-samplingの場合とは逆で、陽性サンプルを増やすことで、不正利用の割合を10%にします。
ランダムフォレストで学習・テストさせると、FNは66から60まで減っていますが、under-samplingと比較すると効果は薄くなっています。 同じ不正利用のサンプルが増えるだけでは、学習モデルに与える影響は小さくとどまる傾向にあるようです。
from imblearn.over_sampling import RandomOverSampler # ランダムにover-sampling ros = RandomOverSampler(ratio={0: X_train.shape[0], 1: X_train.shape[0]//9}, random_state=0) X_train_resampled, y_train_resampled = ros.fit_sample(X_train, y_train) print('X_train_resampled.shape: {}, y_train_resampled: {}'.format(X_train_resampled.shape, y_train_resampled.shape)) print('y_train_resample:\n{}'.format(pd.Series(y_train_resampled).value_counts())) # y_train_resample: # 0 142403 # 1 15822 # dtype: int64 # ランダムフォレストにて学習 rfc = RandomForestClassifier(random_state=0) rfc.fit(X_train_resampled, y_train_resampled) # 検証 print('Train score: {:.4f}'.format(rfc.score(X_train_resampled, y_train_resampled))) print('Test score: {:.4f}'.format(rfc.score(X_test, y_test))) print('Confusion matrix:\n{}'.format(confusion_matrix(y_test, rfc.predict(X_test)))) print('f1 score: {:.4f}'.format(f1_score(y_test, rfc.predict(X_test)))) # Train score: 1.0000 # Test score: 0.9995 # Confusion matrix: # [[142147 14] # [ 60 183]] # f1 score: 0.8318
under-samplingとover-sampling(SMOTE)の組み合わせ
最後に2つを組み合わせてみます。
imbalanced-learnではランダム以外にもいくつかover-samplingの実装が用意されており、その1つにSMOTE(Synthetic Minority Over-sampling Technique)があります。 SMOTEは、今あるサンプルをコピーするのではなく、異なる値を持つサンプルを新たに生成することで増やす方法です。 (陽性サンプル間を結ぶ直線上に新しいサンプルをプロットするイメージのようです。)
まずは、under-samplingで不正利用の割合を1%まで増やし、その後SMOTEで不正利用のサンプルを10倍にすることで不正利用の割合を約10%となるようにします。
学習・テストの結果、FNをRandomUnderSampler単体の場合と同じ43まで減らしている一方で、FPの増加は72まで抑えています。 under-samplingだけでは不正利用のサンプルに寄った過学習を起こしていましたが、SMOTEと組み合わせることで軽減できることを確認しました。
from imblearn.over_sampling import SMOTE # under-samplingで不正利用の割合を1%まで増やす positive_count_train = y_train['Class'].sum() rus = RandomUnderSampler(ratio={0:positive_count_train*99, 1:positive_count_train}, random_state=0) X_train_undersampled, y_train_undersampled = rus.fit_sample(X_train, y_train) print('y_train_undersample:\n{}'.format(pd.Series(y_train_undersampled).value_counts())) # y_train_undersample: # 0 24651 # 1 249 # dtype: int64 # SMOTEで不正利用の割合を約10%まで増やす smote = SMOTEENN(ratio={0:positive_count_train*99, 1:positive_count_train*10}, random_state=0) X_train_resampled, y_train_resampled = smote.fit_sample(X_train_undersampled, y_train_undersampled) print('y_train_resample:\n{}'.format(pd.Series(y_train_resampled).value_counts())) # y_train_resample: # 0 24651 # 1 2490 # dtype: int64 # 検証 rfc = RandomForestClassifier(random_state=0) rfc.fit(X_train_resampled, y_train_resampled) print('Train score: {:.4f}'.format(rfc.score(X_train_resampled, y_train_resampled))) print('Test score: {:.4f}'.format(rfc.score(X_test, y_test))) print('Confusion matrix:\n{}'.format(confusion_matrix(y_test, rfc.predict(X_test)))) print('f1 score: {:.4f}'.format(f1_score(y_test, rfc.predict(X_test)))) # Train score: 0.9996 # Test score: 0.9992 # Confusion matrix: # [[142089 72] # [ 43 200]] # f1 score: 0.7767