前回から引き続き、データ解析のための統計モデリング入門(通称、緑本)を読み進めています。
述べられている理論を整理しつつ、Rでの実装をPythonに置き換えた際のポイントなども深掘りしていきます。
今回は第4章です。実装は以下で公開しています。
4 GLMのモデル選択 -AICとモデルの予測の良さ-
モデルが複雑になるほど、観測データに対する当てはまりは良くなってしまうため、「当てはまりの良いモデル=良いモデル」ではない。
「良い予測を行うモデル=良いモデル」という前提のもと、AICではこの「予測の良さ」を測る。
第3章で扱われている架空植物100個体の種子数のデータを使います。以下の著者サイトから入手できます。目的変数は種子数(y)です。
http://hosho.ees.hokudai.ac.jp/~kubo/ce/IwanamiBook.html
この投稿では種子数データと呼ぶことにします。
4.1 データはひとつ、モデルはたくさん
種子数を予測するモデルを検討する。
前章で現れた3つのモデルに、一定モデル( )を加えた4つのモデルについて考える。
- 体サイズ(
)が影響するxモデル(xモデル)
- 施肥処理(
)が影響するyモデル(yモデル)
- 体サイズと施肥処理の両方が影響するx+fモデル
- 体サイズも施肥処理も影響しない一定モデル
xモデルで推定した結果は以下となります。第3章で得られたものと同じです。
results = sm.GLM(data['y'], sm.add_constant(data['x']), family=sm.families.Poisson()).fit()

4.2 統計モデルの当てはまりの悪さ:逸脱度
これまで、当てはまりの良さを対数尤度 で測ってきました。最も当てはまりの良いモデルは最大対数尤度として、
とします。
この最大対数尤度を使い、当てはまりの悪さを逸脱度Dとして定義する(-2をかけただけです)。
ポアソン分布モデルで可能な最小逸脱度から、モデルの逸脱度Dを引いたものを、そのモデルの残差逸脱度とする。
ポアソン分布モデルで可能な最小逸脱度は、 なので
、
なので
・・・と全データを使ってあてはめた、つまり
となるモデルの逸脱度となります。この比較用の統計モデルは(Rでは)フルモデルと呼ばれます。フルモデルの逸脱度D*は以下で計算されます。
フルモデルの最大対数尤度は-192.89、逸脱度は385.78となります。
xモデルの逸脱度は470.78(-2*-235.39)であることから、残差逸脱度は85.0(470.78-385.78)と計算されます(上の結果の"Deviance"と値が一致していることがわかります)。
# フルモデルの最大対数尤度 full_ll = sum([poisson.logpmf(y_i, y_i) for y_i in data['y']]) # フルモデルの逸脱度(最小逸脱度) full_deviance = (-2) * full_ll
この残差逸脱度が最大になるモデル(=最もあてはまりの悪いモデル)もあります。切片 のみの一定モデルがこれに該当し、Rではnull modelと呼ばれます。
この時の残差逸脱度は89.5となります。
results = sm.GLM(data['y'], np.ones(len(data)), family=sm.families.Poisson()).fit()

4.3 モデル選択規準 AIC
「予測の良さ」を重視したモデルの選択規準としてAICを導入する。
最尤推定するパラメータの個数をkとするとAICは以下で計算されます。 このAICが小さいモデルが良いモデルとなるため、最小のモデルを選択することになります。
残差逸脱度(あてはまりの悪さ)+パラメータ数×2なので、直感的には2kがモデルの複雑度に対するペナルティとなっているように見えます。
4.5 なぜAICでモデル選択してよいのか?
予測の良さを表す平均対数尤度を導入する。そして、最大対数尤度と平均対数尤度の差であるバイアスが、パラメータの数と一致することを示すことで、AICによって予測の良さを計測できることを説明する。
まずは真のモデルを知っている状態からスタートするために、 のポアソン分布モデルから生成される50個のデータを生成します。
np.random.seed(10) # 一定の値になるようにシードを0で固定 # xに4〜12のポアソン乱数をセット data_random = pd.DataFrame(np.random.poisson(8, 50), columns=['y'])
この時の平均値は7.44となっており、真のモデルから少し外れていることがわかります。

この50個のデータを観測されたデータとして、一定モデルでの最尤推定を行います。
切片2.0069( )で、その時の最大対数尤度は-116.47となります。
model = smf.poisson('y ~ 1', data=data_random)
results = model.fit()

最大対数尤度は、たまたま得られた観測データに対するあてはまりの良さでした。
予測の良さ、つまり他のデータにも適用できるかどうかを調べたいのですが、今回は真のモデルを知っているため、この真のモデルから新たにデータ(ここでは50データ×200セット)を作り出します。
そして上で得られたモデルを使って各セットの対数尤度を計算し、その平均値を求めます。これを平均対数尤度( と表記)と呼びます。
平均対数尤度を計算すると-122.48となり、最大対数尤度と比較すると約6小さい(=あてはまりが悪い)ことがわかります。
# 真のモデルから200セットのデータを生成 data_random_200 = [pd.DataFrame(np.random.poisson(8, len(data_random)), columns=['y']) for i in range(200)] # 200セットのデータに対して、β1=2.0069で200個の対数尤度を計算 lls = [] for d in data_random_200: lls.append(sum(poisson.logpmf(d, math.exp(results.params['Intercept'])))) # 平均対数尤度を計算 elogl = np.sum(lls) / len(lls)
「1セットで最大対数尤度を求め、そのモデルを使って200セットの平均対数尤度を求める」というのを200回繰り返してプロットした結果が、以下です。概ね、平均対数尤度(青▲)が最大対数尤度(赤●)よりもあてはまりが悪いことがわかります(ただし、常に平均対数尤度<最大対数尤度とも限らず、ばらつきは大きいものになってます)。
最大対数尤度と平均対数尤度の差をバイアスと呼びます。今回の例では、200回の平均バイアスは1.25となりました。

一般的に真のモデルは不明です。つまり は未知の値となるはずです。
しかし、最尤推定するパラメータをk個持つモデルの平均対数尤度の推定量は、 log L-kとなることが、解析的かつ一般的に導出されている。これにより、先程導入したバイアスb=パラメータ数kと言えることにも着目してください。一定モデルはk=1のため、今回はE(log L)=log L-1となります。
AICはこれに-2をかけたもので、 です。
平均対数尤度は予測の良さを表すので、AICの値が小さいほど、予測の良いモデルと言えます。
これまでは目的変数yだけのモデルを見てきました。これに説明変数xも導入して、2つのモデルを評価します。y、xのいずれも乱数で生成され、2つの変数の間に何の関係も無いデータとします(xは予測の役に立たない変数のはずです)。
: 一定モデル(k=1)
: xモデル(k=2)
まずはデータ200セットで最大対数尤度を比較します。
以下のグラフは(xモデルの最大対数尤度 - 一定モデルの最大対数尤度)の分布ですが、xモデルの方が平均すると0.46大きくなっています。関係ないはずのxを使った何らかのあてはめの方が最大対数尤度は良くなることが確認できます。

一方で、平均対数尤度の差(xモデルの平均対数尤度 - 一定モデルの平均対数尤度)を比較すると、今度は逆に一定モデルの方が平均0.48大きくなります。

したがって、バイアスの差(最大対数尤度の差 - 平均対数尤度の差)は平均0.95で、パラメータ数の差(2-1=1)に近い値となっていることがわかります。分布でもそれほどばらつきが大きくなっていません。
今回の例のように、ネストしたモデル(xモデルが一定モデルの変数 も包含している)の場合、バイアスもばらつきが小さくなります。

200セットの内の1つ目のデータでAICを比較すると、一定モデル:258.1 < xモデル:259.0で、一定モデルを選択すべきとなります(逆に、説明変数xを追加したことで最大対数尤度が1以上増加した場合、xモデルを選択すべきと判定されます)。
欠席数データに対してAICでモデルを選択する
最後に、以前の投稿でも使った欠席数のデータで、AICによるモデル選択を見ていきます。
このデータはUCIで提供されているStudent Performance Data Setです。目的変数は欠席数(absences)です(これまで同様、外れ値の除去や、欠席2回を1回とカウントするなど、若干の前処理を行っています)。
https://archive.ics.uci.edu/ml/datasets/student+performance

ここでは2つのモデルを比較します。
- 年齢(age)が欠席数に影響を与えるとするageモデル
- どの変数も欠席数に影響を与えないとする一定モデル
# ageモデルを作る age_model = smf.poisson('absences ~ age', data=student_data)

# 一定モデルを作る const_model = smf.poisson('absences ~ 1', data=student_data)

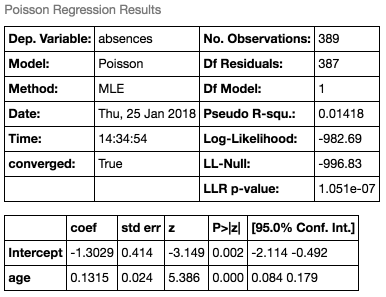
一定モデルの最大対数尤度-996.8に対して、ageモデル-982.7で、約5.9改善されていることがわかります。
次に、フルモデルを求めます。最大対数尤度は-392.9(最小逸脱度は786.7)となります。
# フルモデルの最大対数尤度 full_model_ll = sum([poisson.logpmf(y_i, y_i) for y_i in student_data['absences']]) # -392.854706449 # フルモデルの逸脱度(最小逸脱度) full_model_deviance = (-2) * full_model_ll # 785.709412899
最後に、フルモデルとの残差逸脱度を計算します。
# 一定モデルの残差逸脱度 const_model_d = -2 * (const_model_results.llf - full_model_ll) # 1207.95059528 # ageモデルの残差逸脱度 age_model_d = -2 * (age_model_results.llf - full_model_ll) # 1179.67358728
AICはそれぞれ一定モデルで1210.0(=1208.0+2)、ageモデルで1183.7(=1179.7+4)となり、ageモデルを選択すべき、という結論になります。