データ解析のための統計モデリング入門(通称、緑本)を読み進めています。
述べられている理論を整理しつつ、Rでの実装をPythonに置き換えた際のポイントなども深掘りしていきます。
今回は第7章です。実装は以下で公開しています。
7 一般化線形混合モデル(GLMM) -個体差のモデリング-
GLMでは、説明変数が同じであれば、それ以外の要素は全て均質(=1つの平均λに定まる)という前提を置いている。
しかし、現実にはそうではなく、定量化できていない(原因不明の)個体間の差異がある。
この個体間のばらつきを、データのばらつきとは別の確率分布で表現するようにGLMを拡張したモデルが、一般化線形混合モデル(GLMM)です。
7.1 例題:GLMでは説明できないカウントデータ
架空植物100個体について葉数x(2〜6枚)から種子数y(0〜8個)を予測するモデルを想定する。
- 応答変数yが上限のあるカウントデータなので、第6章で扱った二項分布に従うと仮定する
- 線形予測子
、ロジットリンク関数で最尤推定する
しかし、現実のデータはきれいな二項分布に従うとは限らない。x=4の時の分布(個体数20)を見てみると、谷のような形になることもありうる。
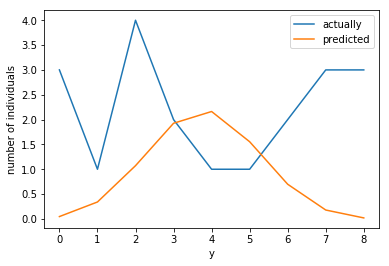
7.2 過分散と個体差
現実のデータは上のように、確率分布が仮定しているよりも、分散が大きくなる(過分散)の傾向が見られる。
x=4において、平均4.05であるのに対して、分散8.37となっており、二項分布が期待する分散2(=8×(4.05/8)×(1-(4.05/8)))よりもかなり大きい。
data4 = data[(data['x']==4)] print('平均:', data4['y'].mean()) # 4.05 print('分散:', data4['y'].var()) # 8.37
データとして識別も定量化もされていない個体差があるため、こうした過分散が起こります。
しかし、個体差を全て特定し、定量化することは不可能です。そのため、これら原因不明の個体差の及ぼす影響を取り込んだ統計モデルが必要です。
7.3 一般化線形混合モデル
個体差の効果をGLMに取り込んだモデルが、一般化線形混合モデル(GLMM)です。
上の線形予測子に、個体iの個体差を表現するパラメータ (-∞〜+∞)を追加する(生存確率は
では高く、
では低く補正されます)。
個体差 などのランダム効果(random effects)を線形予測子に含むため、"混合"と呼ばれます(
や
は固定効果(fixed effects)と言います)。
$$ logit(q_i)=\beta_1+\beta_2 x_i+r_i $$
GLMMでは個体差 はある確率分布に従うと仮定します。
ここでは平均0・標準偏差sの正規分布に従い、また、個体間で相互に独立した確率変数(独立同分布に従う)であると仮定します。確率密度関数 は以下のようになります。
$$ p(r_i \mid s)=\frac{1}{\sqrt{2 \pi s^2}}\exp(-\frac{r_i^2}{2s^2}) $$
rが小さいほど均質な集団、大きくなるほど個体差が大きい集団というふうになります。
個体差のみから確率 を計算するモデル(下式)で、50個体の生存種子数をシミュレーションした図を示します。
s=0.5(青)の時の標本分散1.04だったのに対して、s=3.0(赤)では9.72となり、分布がかなりばらついていることがわかります。
$$ q_i = \frac{1}{1+\exp(-r_i)} $$
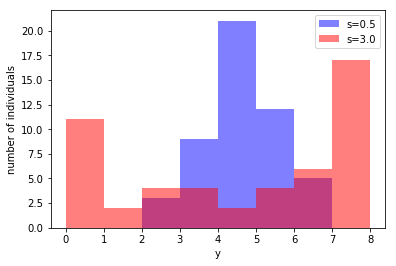
7.4 一般化線形混合モデルの最尤推定
個体差 はそのままでは最尤推定できません。
最尤推定して が得られたとしても、100個のデータに対して100個のパラメータを当てはめたフルモデルになるからです。
一方で、 と
を推定するには、
を除いた式にする必要があります。そのために、
で積分することで
を消します。
$$ L_i = \int_{-\infty}^{\infty} p(y_i \mid \beta_1, \beta_2, r_i) p(r_i \mid s) dr_i $$
式を見ると、左の項( )は二項分布、右の項(
)は正規分布になっています。二項分布の項は
毎に異なるパラメータとなる(つまりサンプルサイズの数だけ二項分布がある)ため、かけあわせることで過分散な確率分布を作り出すことができる。
全データの尤度は100個体分の積となります。 はもうありません。最尤推定では
を計算します。
$$ L(\beta_1, \beta_2, s)=\prod_i L_i $$
PypeRでPythonからRを実行する
さて、RではglmmMLパッケージで簡単にGLMMの最尤推定ができます。
しかし、PythonではGLMMで最尤推定を計算するライブラリというのは無く、statsmodelsやPyStanでも実装されていないようです。(さらに複雑化したモデルにも対応できるベイズモデルが既に実装・提供されているからでしょうか。)
そこで、タイトルと異なっていて不本意ではありますが、PythonからRのコードを実行するPypeRを導入します。以降のコードは実行環境にRがインストールされている前提となります。
http://bioinfo.ihb.ac.cn/softwares/PypeR/
まずはpyper.Rオブジェクトを作成します。
import pyper # pip install pyper r = pyper.R() type(r) # pyper.R
得られたRオブジェクトを、r("実行したいRコード")のように関数形式で呼び出すことで、Rのコードが実行されます。
assignメソッドを使うことで、Pythonのオブジェクトを任意の名前でRに渡すこともできます。PandasのDataFrameやSeriesもそのまま渡せます。
r.assign("data", data) print(r("summary(data)")) # try({summary(data)}) # N y x id # Min. :8 Min. :0.00 Min. :2 Min. : 1.00 # 1st Qu.:8 1st Qu.:1.00 1st Qu.:3 1st Qu.: 25.75 # Median :8 Median :3.00 Median :4 Median : 50.50 # Mean :8 Mean :3.81 Mean :4 Mean : 50.50 # 3rd Qu.:8 3rd Qu.:7.00 3rd Qu.:5 3rd Qu.: 75.25 # Max. :8 Max. :8.00 Max. :6 Max. :100.00
それでは、glmmMLパッケージで最尤推定を行います。事前に> install.packages("glmmML")でインストールしておきます。
glmmMLの引数を見ると、GLMで渡したパラメータに加えて、cluster = idが渡されています。clusterは個体ごとに異なるパラメータを期待しており、今回は個体ごとに異なる通し番号を渡しています。
r(""" library(glmmML) result <- glmmML(cbind(y, N-y) ~ x, data = data, family = binomial, cluster = id) """))
推定した結果をPythonのオブジェクトに戻す時は、getメソッドを使います。
result = r.get("result") print(result)
resultオブジェクト(dict)には、以下の値を持っていました。
{'aic': 275.414,
'boot': 0,
'bootP': None,
'call': array(['glmmML', 'cbind(y, N - y) ~ x', 'binomial', 'data', 'id'],
dtype='<U19'),
'cluster.null.deviance': 513.836,
'cluster.null.df': 98,
'coef.sd': array([ 0.878, 0.207]),
'coefficients': array([-4.19 , 1.005]),
'converged': True,
'deviance': 269.414,
'df.residual': 97,
'info': 0,
'posterior.modes': array([-1.337, 0.195, 0.967, 2.007, 0.195, -1.337, -1.337, 3.556,
0.195, 2.962, -1.337, -1.337, -1.337, -1.337, -1.337, 0.195,
1.526, 2.469, 1.526, 3.556, 0.069, -0.648, -1.951, 2.05 ,
-0.648, 0.069, -1.951, -1.951, -1.951, 0.608, 1.546, 1.546,
-1.951, -1.951, -1.951, 3.639, 1.082, 0.069, -1.951, 3.639,
1.142, -0.834, -2.638, 0.624, 1.142, -0.834, 2.884, -0.834,
1.801, 1.801, 2.884, -2.638, 0.157, 1.801, -0.312, -1.507,
-0.312, -0.834, 2.884, -2.638, 0.938, -0.768, -1.742, -1.233,
-1.233, 0.938, 2.177, 0.938, -3.378, 0.938, 2.177, 2.177,
2.177, 0.237, 0.938, -0.768, -0.296, -2.376, 2.177, -1.742,
-3.256, -3.256, -4.158, 1.536, 1.536, 1.536, 1.536, 1.536,
1.536, 1.536, 1.536, -4.158, -0.663, -2.156, -2.156, 0.089,
-1.215, 0.089, -2.653, 1.536]),
'prior': 'gaussian',
'sigma': 2.408,
'sigma.sd': 0.220,
'terms': array(['~', 'cbind(y, N - y)', 'x'],
dtype='<U15'),
'variance': array([[ 7.703e-01, -5.691e-04, -1.456e-03],
[ -5.691e-04, 4.304e-02, 1.181e-02],
[ -1.456e-03, 1.181e-02, 4.849e-02]])}
7.5 現実のデータ解析にはGLMMが必要
GLMとGLMMの使い分けは「同じ個体から採取されているか」にかかっており、「全て異なる個体から採取している(反復と呼ばれます)」場合はGLM、「同じ個体から採取している(疑似反復と呼ばれます)」場合はGLMMを適用すべき。
ある個体から1回しか採取されない場合は、個体差がわからないので変数化する必要はありません。そのため線形予測子は固定効果の変数のみを持ちます。
$$ logit(q_i) = \beta_1 + \beta_2 x_i $$
しかし、同じ個体iから複数回採取する場合は、個体差が識別できるので変数化( )してモデルに加える必要があります。
$$ logit(q_i) = \beta_1 + \beta_2 x_i + r_i $$
さらに各個体が存在する場所jが異なっており、それぞれの場所にある個体から複数回採取されている、といったケースでは、さらにその場所も変数( )として加えます。
$$ logit(q_i) = \beta_1 + \beta_2 x_i + r_i + r_j $$
こうしてランダム効果が複数の変数で表現されると、最尤推定で多重積分する必要があります。このようなときには、代わりに階層ベイズモデルとMCMCが組み合わせた方法が使われます(第8章以降のテーマ)。
